Annotation wizard: Difference between revisions
Jump to navigation
Jump to search
(Created page with "Here's an example of using the annotation wizard: <source lang="python"> import pymol ...") |
No edit summary |
||
| (2 intermediate revisions by the same user not shown) | |||
| Line 1: | Line 1: | ||
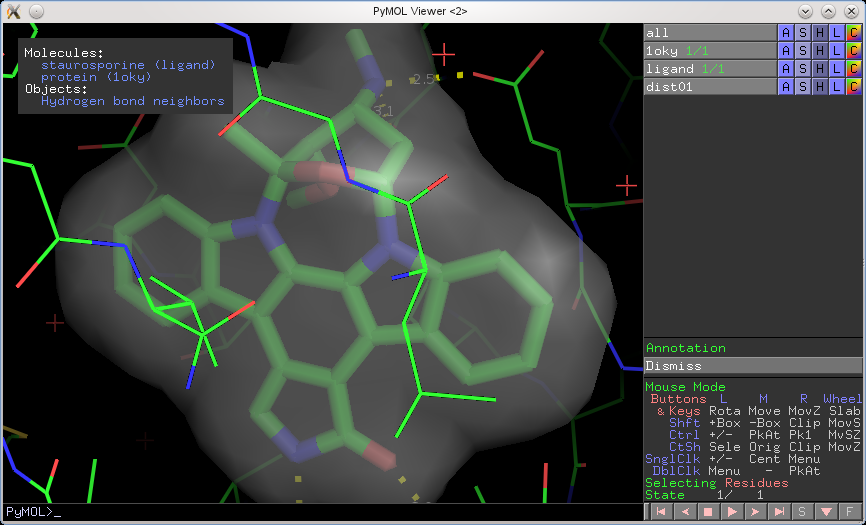
Here's an example of using the annotation wizard: | Here's an example of using the annotation wizard (see top-left corner): | ||
[[Image:Ann.png|450px]] | |||
<source lang="python"> | <source lang="python"> | ||
| Line 23: | Line 25: | ||
pymol.session.annotation["1oky"] = state_dict | pymol.session.annotation["1oky"] = state_dict | ||
</source> | |||
The following source shows you how to use the annotation wizard for multiple objects. | |||
<source lang="python"> | |||
import pymol | |||
# fetch a protein and setup the view | |||
cmd.fetch("1oky", async=0) | |||
cmd.extract("ligand", "resn STU") | |||
cmd.show_as("sticks", "resn STU") | |||
cmd.show("surface", "ligand") | |||
cmd.flag("ignore", "not rep surface") | |||
cmd.set("surface_color", "grey") | |||
cmd.set("transparency", 0.3) | |||
cmd.distance("(ligand)", "(poly)", quiet=1, mode=2, label=1) | |||
# turn on the annotation wizard + prompt | |||
cmd.wizard("annotation") | |||
cmd.set("wizard_prompt_mode", 1) | |||
pymol.session.annotation = {} | |||
# using the annotation wizard for multiple objects: | |||
prot_dict = { 1: ['\\999Protein:',' \\459protein (1oky)',]} | |||
lig_dict = { 1: ['\\999Ligand:',' \\459staurosporine (ligand)',]} | |||
dist_dict = { 1: ['\\999Objects: ',' \\459Hydrogen bond neighbors',]} | |||
pymol.session.annotation["1oky"] = prot_dict | |||
pymol.session.annotation["ligand"] = lig_dict | |||
pymol.session.annotation["dist01"] = dist_dict | |||
</source> | </source> | ||
[[Category:Wizards]] | [[Category:Wizards]] | ||
Latest revision as of 10:23, 30 January 2012
Here's an example of using the annotation wizard (see top-left corner):
import pymol
# fetch a protein and setup the view
cmd.fetch("1oky", async=0)
cmd.extract("ligand", "resn STU")
cmd.show_as("sticks", "resn STU")
cmd.show("surface", "ligand")
cmd.flag("ignore", "not rep surface")
cmd.set("surface_color", "grey")
cmd.set("transparency", 0.3)
cmd.distance("(ligand)", "(poly)", quiet=1, mode=2, label=1)
# turn on the annotation wizard + prompt
cmd.wizard("annotation")
cmd.set("wizard_prompt_mode", 1)
pymol.session.annotation = {}
state_dict = {1: ['\\999Molecules:',' \\459staurosporine (ligand)',' \\459protein (1oky)','\\999Objects:',' \\459Hydrogen bond neighbors',]}
pymol.session.annotation["1oky"] = state_dict
The following source shows you how to use the annotation wizard for multiple objects.
import pymol
# fetch a protein and setup the view
cmd.fetch("1oky", async=0)
cmd.extract("ligand", "resn STU")
cmd.show_as("sticks", "resn STU")
cmd.show("surface", "ligand")
cmd.flag("ignore", "not rep surface")
cmd.set("surface_color", "grey")
cmd.set("transparency", 0.3)
cmd.distance("(ligand)", "(poly)", quiet=1, mode=2, label=1)
# turn on the annotation wizard + prompt
cmd.wizard("annotation")
cmd.set("wizard_prompt_mode", 1)
pymol.session.annotation = {}
# using the annotation wizard for multiple objects:
prot_dict = { 1: ['\\999Protein:',' \\459protein (1oky)',]}
lig_dict = { 1: ['\\999Ligand:',' \\459staurosporine (ligand)',]}
dist_dict = { 1: ['\\999Objects: ',' \\459Hydrogen bond neighbors',]}
pymol.session.annotation["1oky"] = prot_dict
pymol.session.annotation["ligand"] = lig_dict
pymol.session.annotation["dist01"] = dist_dict